Metody sekwencjonowania pojedynczych komórek to przełomowe technologie umożliwiające badanie i charakterystykę poszczególnych komórek w heterogenicznych tkankach nowotworowych. W przypadku raka pomaga nam to zrozumieć, dlaczego dla niektórych typów nowotworów trudno jest opracować skuteczną terapię. Podczas gdy genom (DNA) i transkryptom (RNA) można obecnie rutynowo badać na poziomie pojedynczej komórki, profilowanie proteomu (białek) w pojedynczych komórkach jest znacznie trudniejsze, ponieważ obecne metody nie są wystarczająco czułe. Ze względu na to, że białka wykonują wiele ważnych funkcji w komórce i ulegają modyfikacjom pod wpływem zmian fizjologicznych, np. stanów chorobowych, brak takiej technologii znacznie ogranicza nasze zrozumienie biologii tkanek.
– Proteomika jest obecnie zdominowana przez metody oparte o spektrometrię mas – mówi dr Javier Alfaro z Międzynarodowego Centrum Badań nad Szczepionkami Przeciwnowotworowymi w Gdańsku. Niestety takie urządzenia badawcze są drogie i słabo sekwencjonują białka w złożonych mieszaninach. – Nie identyfikujemy każdego aminokwasu w sekwencji, tak jak robimy to dla DNA w złożonych mieszaninach – kontynuuje dr Javier Alfaro. W tym przypadku postęp wymaga transformacji technologicznej, podobnej do tej, która zrewolucjonizowała sekwencjonowanie DNA, ale z zastosowaniem do analizy białek. – Problem polega na tym, że w przeciwieństwie do DNA, nie mamy sposobu na amplifikację (namnażanie) białek – a to stanowi duże wyzwanie dla czułości obecnego oprzyrządowania – stwierdza dr Javier Alfaro.
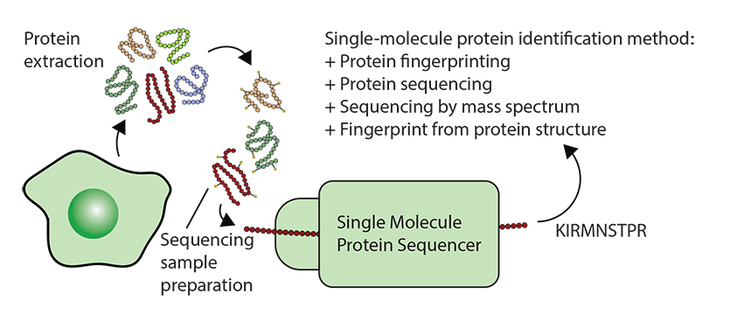
Nowoczesne metody sekwencjonowania mogą pewnego dnia znaleźć zastosowanie w sekwencjonowaniu profili białkowych w pojedynczej komórce lub być podstawą nowych, bardziej czułych technik diagnostycznych. Według standardowego protokołu po ekstrakcji białek z komórki następuje przygotowanie próbki specyficzne dla danej techniki sekwencjonowania. Każda z technologii identyfikuje białko w inny sposób, jak wskazano na grafice.
Sekwencjonowanie białek na poziomie pojedynczej cząsteczki pozwoli na badanie białek na poziomie pojedynczej komórki, nawet przy niewielkiej ilości białka. Takie techniki umożliwiłyby rutynowe profilowanie proteomu, podobnie do obecnie stosowanego sekwencjonowania RNA pojedynczych komórek. Umożliwi to poznanie składu białek pojedynczych komórek i potencjalnie – prowadzenie medycznych badań diagnostycznych i przesiewowych w czasie rzeczywistym. – Nowe technologie wymagają opracowania nowych sposobów chemicznego i biologicznego znakowania, które pozwolą nam wykrywać za pomocą fluorescencji nie tylko określone aminokwasy w białkach, ale również ich modyfikacje – zwraca uwagę dr Adam Pomorski z Uniwersytetu Wrocławskiego.
Podczas pisania pracy wielu znanych biofizyków, technologów, klinicystów i bioinformatyków zostało zaproszonych do podzielenia się swoimi perspektywami jako równorzędni współpracownicy. – W dzisiejszych czasach wszyscy jesteśmy mistrzami w swoich specyficznych dziedzinach. Współpraca specjalistów, jako równych sobie, pozwala połączyć ich energię i różnorodne perspektywy, co jest niezbędne do przełamywania barier i uzyskania unikalnego efektu naukowego – konkluduje dr Javier Alfaro.
Uniwersytet Gdański
























KOMENTARZE