Na przestrzeni dziejów ludzie zawsze byli narażeni na choroby zakaźne wywoływane przez bakterie i wirusy. W ciągu ostatnich 18 lat na całym świecie znacząco wzrosła śmiertelność z powodu ostrych zespołów oddechowych wywołanych przez koronawirusy, w tym SARS i MERS. Wraz z obecnie trwającą pandemią COVID-19, która pochłonęła już ponad milion istnień ludzkich, mamy pilną potrzebę opracowania nowych sposobów zwalczania zakażeń wywołanych koronawirusami.
Choroba COVID-19 jest wywoływana przez SARS-CoV-2, betakoronawirusa, którego genom stanowi pojedyncza bardzo długa cząsteczka RNA, kodująca białka wirusowe. Koronawirusy są rekordzistami wśród wirusów pod względem długości RNA – ich genom ma około 30 000 nukleotydów. Podobnie jak w przypadku innych wirusów, RNA wirusa SARS-CoV-2 tworzy struktury przestrzenne, które odgrywają kluczową rolę w regulowaniu mechanizmu działania wirusa, w tym jak wirus replikuje się w komórkach ludzkich. Do tej pory przebadano jedynie kilka pojedynczych elementów strukturalnych w RNA koronawirusów.
Naukowcy z IIMCB razem ze współpracownikami z Uniwersytetów w Groningen i w Lejdzie zbadali struktury tworzone przez materiał genetyczny wirusa SARS-CoV-2, wykorzystując szereg zaawansowanych technik. Prace badawcze koordynowane przez dr. Danny'ego Incarnato z Uniwersytetu w Groningen obejmowały zbadanie struktury RNA koronawirusa SARS-CoV-2 w celu uzyskania informacji o wzajemnych oddziaływaniach wszystkich zasad w genomie. Cząsteczki wirusowego RNA zostały zbadane doświadczalnie zarówno bezpośrednio w zainfekowanych komórkach ludzkich, jak i po wyizolowaniu z komórek. Następnie badacze zastosowali komputerowe metody analizy danych, aby zidentyfikować takie regiony wirusowego RNA, które tworzą stabilne struktury przestrzenne. W ten sposób po raz pierwszy określono strukturę całego RNA koronawirusa. Wśród 87 najsilniej ustrukturalizowanych regionów, ok 10% wykazuje wysokie podobieństwo pomiędzy różnymi koronawirusami, co sugeruje, że mogą mieć specjalne znaczenie.
– Najpierw zidentyfikowaliśmy struktury in vitro, a następnie potwierdziliśmy ich obecność w RNA wirusów wewnątrz zainfekowanych komórek. Oznacza to, że otrzymane przez nas wyniki są bardzo solidne. Ponadto, w niektórych strukturach RNA zidentyfikowane zostały motywy strukturalne, które mogą być wykorzystane jako cele działania cząsteczek chemicznych, potencjalnie zaburzających funkcjonowanie wirusowego RNA. Co więcej, szereg struktur jest zachowanych ewolucyjnie pomiędzy różnymi koronawirusami, co oznacza, że skuteczny lek ukierunkowany na SARS-CoV-2 mógłby być efektywny również w przyszłości przeciwko nowym szczepom wirusa – wyjaśnia Incarnato.
Naukowcy zidentyfikowali również fragmenty RNA SARS-CoV-2, które są wewnętrznie nieustrukturyzowane. – Mogą one stanowić cel działania terapii antysensownymi oligonukleotydami – tłumaczy dalej badacz. Dodając krótkie nici kwasu nukleinowego, które mogą sparować się z nieustrukturyzowanymi sekcjami wirusowego RNA, można utworzyć struktury dwuniciowe, stanowiące cel działania naturalnych mechanizmów molekularnych, jakimi komórki bronią się przed obcymi RNA. Podsumowując, przedstawione wyniki badań stanowią fundament dla przyszłych prac, mających na celu opracowanie potencjalnych leków do leczenia zakażeń wywołanych zarówno przez SARS-CoV-2, jak i inne koronawirusy.
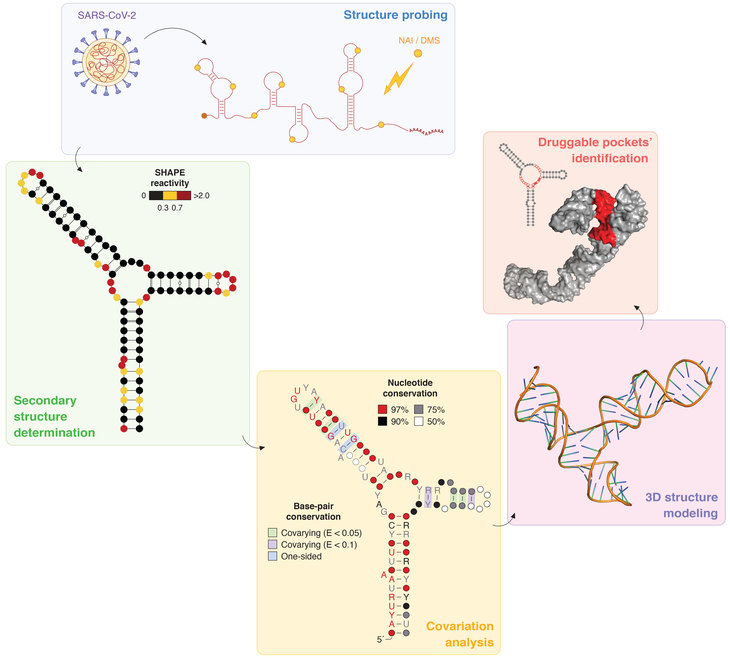
Schematyczne przedstawienie podejścia doświadczalnego przyjętego przez naukowców. Po przeprowadzeniu „próbkowania” struktury genomu RNA SARS-CoV-2 przy użyciu określonych związków chemicznych, ustalono, które zasady tworzą ze sobą pary i w ten sposób określono drugorzędową strukturę RNA całego genomu wirusa. Na podstawie tych informacji zostały opracowane modele struktury przestrzennej najbardziej stabilnych elementów struktury RNA, co pozwoliło na identyfikację miejsc najbardziej podatnych na odziaływanie z małymi cząsteczkami chemicznymi, które mogą stanowić cele dla opracowania nowych przeciwwirusowych strategii terapeutycznych. W ostatecznej analizie strukturalnej badacze skupili się na tych elementach struktury RNA, które okazały się silnie zachowane ewolucyjnie pomiędzy SARS-CoV-2 i innymi koronawirusami. (ryc. Danny Incarnato)
– Ta praca nie byłaby możliwa bez współpracy międzynarodowych zespołów badaczy z Holandii i Polski. Razem opracowaliśmy nowy sposób wyszukiwania potencjalnych słabych punktów w dużych wirusowych RNA. Wyniki naszej wspólnej pracy stanowią istotną podstawę do opracowania innowacyjnych strategii terapeutycznych ukierunkowanych na RNA, w walce z infekcjami SARS-CoV-2 – mówi Janusz Bujnicki, kierownik Laboratorium Bioinformatyki i Inżynierii Białka w Międzynarodowym Instytucie Biologii Molekularnej i Komórkowej w Warszawie.
Artykuł opisujący wyniki prac nad strukturą RNA SARS-CoV-2 został opublikowany w czasopiśmie „Nucleic Acids Research”, a do tego wyróżniony jako „przełomowy” przez oceniających go recenzentów. Pełna treść artykułu „Genome-wide mapping of therapeutically-relevant SARS-CoV-2 RNA structures” znajduje się tutaj.























KOMENTARZE