Predykcja specyficznych epitopów w obrębie immunogennych białek prowadzona jest w oparciu o analizy bioinformatyczne, wykorzystujące takie właściwości jak: hydrofobowość, lokalizację analizowanej sekwencji w obrębie białka czy właściwości fizyczno-chemiczne poszczególnych aminokwasów, tworzących daną sekwencję [8-11]. Następnie wyniki uzyskane metodą in silico weryfikowane są przy wykorzystaniu odpowiednich technik laboratoryjnych, prowadzących do powstania syntetycznych peptydów o znanej sekwencji aminokwasowej [12, 13].
W przyrodzie występuje 20 aminokwasów stanowiących podstawowy budulec wszystkich białek, a co za tym idzie – także peptydów. W badaniach laboratoryjnych wykorzystywane są syntetyczne aminokwasy, które różnią się od tych występujących w naturze obecnością zabezpieczających grup bocznych. Ich rolą jest zapobieganie powstawaniu nieliniowych produktów syntezy. Wśród najczęściej wykorzystywanych grup zabezpieczających wymienia się Fmoc (ang. fluorenylmethyloxycarbonyl protecting group) oraz tBu/tBoc (ang. tertbutyloxycarbonyl protecting group) i analogicznie – najczęściej stosowaną strategią syntezy jest strategia Fmoc/tBu [14].
Metoda klasyczna
Chemiczna synteza peptydów, w tym także epitopów, prowadzona może być w dwojaki sposób. Pierwsza metoda wykorzystuje syntezę w roztworze (metoda klasyczna), natomiast druga technika opiera się na syntezie na nośniku stałym, a kluczowa różnica pomiędzy nimi związana jest ze środowiskiem, w jakim znajduje się grupa zabezpieczająca grupę karboksylową pierwszego aminokwasu w sekwencji [15, 16].
Metoda Pepscan
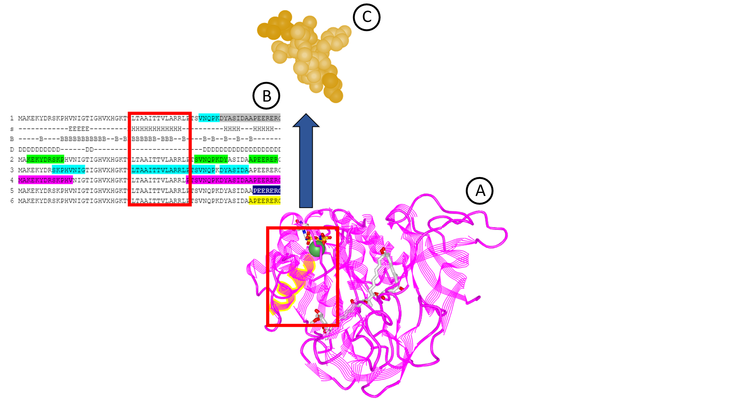
Przykładem syntezy w fazie stałej jest metoda Pepscan, która umożliwia jednoczesne mapowanie nawet kilkudziesięciu epitopów [17]. Przyłączanie kolejnych aminokwasów prowadzone jest na polietylenowych nośnikach (pinach), podczas gdy mieszanina reagentów umieszczana jest w dołkach płytki wielodołkowej, w których następnie umieszczane są poszczególne piny. Uzyskane w ten sposób nawet kilkunastoaminokwasowe peptydy mogą być badane pod kątem ich immunoreaktywności i specyficzności w obecności przeciwciał [12, 13].
Zastosowanie metody Pepscan umożliwiło naukowcom z Katedry Mikrobiologii UJ CM w Krakowie oraz badaczom z IITD PAN we Wrocławiu przebadanie blisko 400 sekwencji aminokwasowych, pochodzących z czterech immunogennych białek Streptococcus agalactiae (ang. group B Streptococcus, GBS), takich jak: enolaza, czynnik elongacji Tu (EF-Tu), dehydrogenaza 5’-monofosforanu inozyny (IMPDH) oraz białko opiekuńcze GroEL [18].
Spośród tych kilkuset badanych sekwencji do dalszych badań wybrano 11 wysoce reaktywnych i specyficznych peptydów (rdzennych i modyfikowanych), a uzyskane wyniki stanowiły podstawę do dalszych badań nad nowym testem immunoenzymatycznym, służącym do diagnostyki zakażeń i nosicielstwa GBS wśród kobiet ciężarnych, który opracowywany jest w ramach grantu TANGO 2 nr TANGO2/340018/NCBR/2017 pt. „Innowacyjny test diagnostyczny do wykrywania zakażeń Streptococcus agalactiae i nosicielstwa wśród ciężarnych”, finansowanego ze środków Narodowego Centrum Nauki i Narodowego Centrum Badań i Rozwoju o wartości 1 100 000,00 zł.
Niniejszy test może stanowić obiecującą alternatywę lub uzupełnienie dla obecnie wykorzystywanych klasycznych metod hodowlanych stosowanych w diagnostyce nosicielstwa GBS wśród kobiet ciężarnych, których istotnym ograniczeniem jest długi czas oczekiwania na wynik. Ponadto istnieje możliwość wykorzystania immunoreaktywnych epitopów jako składowych nowej podjednostkowej szczepionki przeciwko zakażeniom GBS u noworodków.

Autorki:
Anna Dobrut, Zakład Molekularnej Mikrobiologii Medycznej, Katedra Mikrobiologii, Uniwersytet Jagielloński Collegium Medicum, Kraków
Monika Brzychczy-Włoch, Zakład Molekularnej Mikrobiologii Medycznej, Katedra Mikrobiologii, Wydział Lekarski, Uniwersytet Jagielloński Collegium Medicum, Kraków
























KOMENTARZE