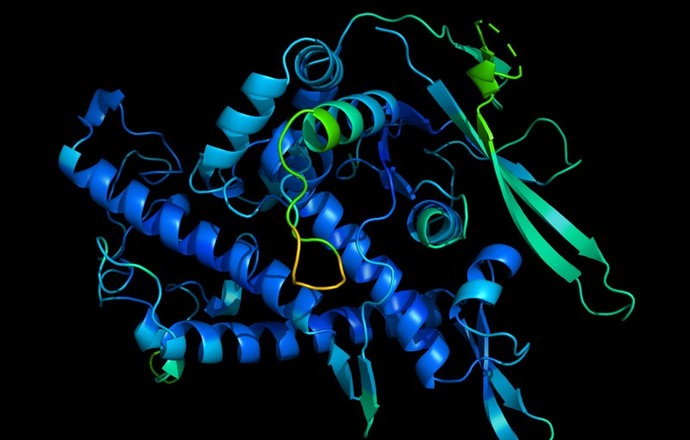
cellPACK to narzędzie wykorzystujące dane biologiczne do tworzenia modeli 3D struktur komórkowych, złożonych z milionów cząsteczek składowych. Dodatkowo, oferowany pakiet umożliwia przechowywanie, wizualizację, analizę i interakcje z wynikami innych modeli, które są dostępne dla różnych odbiorców. cellPACK stanowi bazę danych poszczególnych cząsteczek mezoskali oraz przykładów modeli strukturalnych dla każdego z nich.
– Zakres mezoskali obejmuje struktury biologiczne, które wahają się od 10 do 100 nanometrów – tłumaczy prof. Arthur Olson z Zakładu Zintegrowanej Biologii Doświadczalnej i Obliczeniowej, Scripps Research Institute, San Diego. – Do struktur mieszczących się w tym zakresie można zaliczyć: wirusy, organelle komórkowe, duże kompleksy molekularne i inne wewnątrzkomórkowe składniki środowiska komórkowego.
Narzędzie to ma dodatkowe systemy umożliwiające przestrzenne upakowanie cząsteczek wewnątrz komórki do stworzenia optymalnego modelu. Poszczególne składniki są w stochastyczny sposób pakowane w specyficzne struktury zapewniające przestrzenne ułożenie. Każdy ze składników ma określenie jego właściwości oraz możliwości oddziaływania z innymi cząsteczkami. Umożliwia to stworzenie listy potencjalnych połączeń oraz możliwości upakowania w pożądaną strukturę. Do uzyskania tego potrzebna jest głęboka analiza oraz przeprowadzenie wielu symulacji obrazujących potencjalne struktury dla oddziaływania pomiędzy poszczególnymi cząsteczkami składowymi.
- Oprogramowanie wykorzystuje dane strukturalne mezoskalowego środowiska, mogące zawierać struktury atomowe na poziomie pojedynczych cząsteczek biologicznych (białek, DNA, RNA, itp), mikroskopijne struktury molekularne, kształty oraz rozmieszczenie organelli komórkowych, jak i genomowe oraz białkowe dane opisujące poziom ekspresji poszczególnych składników molekularnych i ich względną ilość lub stężenie w środowiskach komórkowych – wyjaśnia prof. Arthur Olson. - cellPACK wykorzystuje następnie wszystkie te informacje do syntezy jednego lub więcej modeli 3D, zgodnych statystycznie z wszystkim dostępnymi informacjami.
Zastosowanie w tym przypadku specyficznych algorytmów usprawnia oraz ułatwia przeprowadzenie procedur powiązanych z modelowaniem oraz wizualizacją. - cellPACK jest wiele tysięcy bardziej wydajny od ręcznego tworzenia modeli – podkreśla prof. Arthur Olson. - Kilka lat temu, zajęło mi parę tygodni stworzenie modelu osocza krwi. cellPACK umożliwia jego stworzenie w kilka sekund, przy zastosowaniu tych samych informacji. Uważam, że tworzenie takich modeli symulacyjnych stanowiło „wąskie gardło” w zakresie symulacji dużych, dynamicznych systemów. cellPACK może usunąć ten efekt, umożliwiając wiele jednoczesnych symulacji z wykorzystaniem statystycznie poprawnych modeli tego samego systemu.
























KOMENTARZE