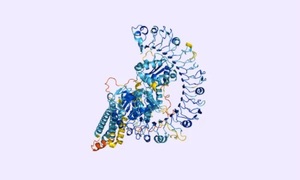
Fot. Potencjalne białko odporności roślin na choroby At1g58602, AlphaFold
– Podpisuję się pod opinią innych naukowców, że jest to wydarzenie tej skali, co poznanie sekwencji genomu człowieka, jeśli nie większe. To jest rewolucja, która zmienia sposób pracy badaczy – ocenia w rozmowie z PAP dr Jan Kosiński z EMBL. Jak wyjaśnia, naukowcy potrzebują informacji o strukturze 3D białek, żeby lepiej zrozumieć mechanizm działania białek w komórkach i całych komórek. – W kontekście medycyny, znając struktury 3D białek, można projektować leki, np. niektóre z cząsteczek, które zaproponowano do zwalczania koronawirusa, zostały zaprojektowane właśnie na podstawie takich struktur – tłumaczy dr Kosiński. Dodaje, że w przypadku mutacji białek prowadzących do rozwoju raka można lepiej zrozumieć, w jaki sposób mutacja zmienia białko, i próbować to naprawić. – To tylko niektóre przykłady, ale jest ich znacznie więcej. To trochę tak, jakbyśmy dali dziecku wszystkie klocki świata, aby mogło budować, co zechce. I to zupełnie za darmo – porównuje.
Genom człowieka zawiera instrukcję do budowy ponad 20 tys. białek, jednakże tylko dla ok. 1/3 tych cząsteczek udało się eksperymentalnie – przy pomocy badań krystalograficznych i mikroskopii krioelektronowej – określić strukturę trójwymiarową. A w wielu wypadkach struktura ta została poznana tylko częściowo. W 2020 r. możliwości przewidywania struktur 3D białek zmieniły się dzięki stworzeniu systemu sztucznej inteligencji AlphaFold, który opracowała firma DeepMind w Londynie. Pozwolił on przewidzieć strukturę niemal całego ludzkiego proteomu, czyli 98,5 proc. z ponad 20 tys. ludzkich białek. Dzięki współpracy DeepMind i EMBL baza danych ze strukturami białek będzie dostępna w sposób otwarty i bezpłatny dla wszystkich. – Eksperymenty mające na celu określenie trójwymiarowej struktury białka mogą trwać wiele miesięcy, a nawet lat. Teraz wystarczy wejść na stronę bazy i ściągnąć plik. To bardzo przyspieszy prace badawcze – zauważa dr Kosiński. W bazie danych – łącznie z proteomem człowieka – znalazło się blisko 350 tys. struktur białek, w tym również białka 20 biologicznie modelowych organizmów, na których prowadzone są niezliczone badania naukowe, takich jak m.in. bakteria Escherichia coli, kukurydza, muszka owocowa, mysz, ryba danio, zarodziec malarii i prątki gruźlicy. Naukowcy podkreślają, że modele białek obecnych w bazie charakteryzują się różną dokładnością, mimo to udostępnienie ich zrewolucjonizuje badania w wielu różnych dziedzinach nauk przyrodniczych. Do końca roku baza powiększy się do 130 mln białek różnych organizmów.
Dr Kosiński zwraca uwagę, że większość ludzkich białek występuje w różnych wersjach. – Tych wszystkich wersji w bazie danych nie ma, ale jeśli naukowiec chce uzyskać trójwymiarową strukturę danej wersji białka, może ściągnąć program AlphaFold i ją poznać – zaznacza. DeepMind udostępnił bowiem ulepszoną, najnowszą wersję tego systemu bezpłatnie. Metodologia stojąca za najnowszą, wysoce innowacyjną wersją AlphaFold oraz jego otwarty kod źródłowy zostały opublikowane w jednym z ostatnich numerów czasopisma „Nature”. Dzięki temu naukowcy mogą uzyskać model trójwymiarowej struktury białka nawet w ciągu godzin lub minut. Jak wyjaśnia dr Kosiński, system AlphaFold używa technik, które zaprojektowano do tłumaczenia tekstów. – Białka to łańcuchy aminokwasów, które zwijają się w różne kształty. AlphaFold „tłumaczy” te sekwencje aminokwasów w trójwymiarową strukturę białka – obrazuje badacz.
Skuteczność tego programu została potwierdzona pod koniec 2020 r. w olimpiadzie przewidywania struktur 3D białek (eksperyment CASP – Critical Assessment of protein Structure Prediction), która odbywa się co dwa lata. Okazało się, że modele białek wysłane przez firmę DeepMind, która stworzyła system, były dużo lepsze, niż te uzyskane z użyciem innych metod. AlphaFold pozwala też ocenić poziom jakości przewidywanej struktury 3D białka. – Chcemy dać naukowcom korzystającym z bazy naprawdę jasny sygnał, na których częściach przewidywanej struktury mogą polegać – komentuje dla serwisu Nature.com Kathryn Tunyasuvunakool, pierwsza autorka pracy opublikowanej na łamach „Nature”, w której opisano przewidywania struktur ludzkich białek.
W miarę identyfikowania nowych białek i zwiększenia dokładności przewidywań ich struktury 3D baza udostępniona przez EMBL i DeepMind będzie aktualizowana. – To będzie jeden z najważniejszych zbiorów danych od czasu mapowania ludzkiego genomu – akcentuje Ewan Birney, zastępca dyrektora generalnego EMBL i dyrektor EMBL-EBI, cytowany w informacji prasowej przesłanej PAP. Jego zdaniem udostępnienie struktur AlphaFold międzynarodowej społeczności naukowej otwiera wiele nowych dróg badawczych dotyczących m.in. chorób zaniedbanych czy nowych enzymów dla biotechnologii. – Jest to wspaniałe, nowe narzędzie naukowe, które uzupełnia istniejące technologie i pozwoli nam przesunąć granice naszego rozumienia świata – podsumowuje Birney.
AlphaFold jest już wykorzystywany w badaniach przez różne instytucje naukowe. Centre for Enzyme Innovation (CEI) University of Portsmouth w Wielkiej Brytanii chociażby wykorzystuje go do opracowania szybszych enzymów do recyklingu niektórych tworzyw sztucznych jednorazowego użytku, które najbardziej zanieczyszczają ziemię. Zespół z Uniwersytetu Kolorado w Boulder wykorzystuje przewidywania AlphaFold do badania oporności bakterii na antybiotyki, podczas gdy grupa z Uniwersytetu Kalifornijskiego w San Francisco – do lepszego zrozumienia biologii wirusa SARS-CoV-2.
PAP




















KOMENTARZE